Mit Mathe durch das Netz der Algen und Bakterien
Wissenschaftler schlagen Brücken zwischen Mathematik und Biologie
22.10.2012
Wissenschaftler schlagen Brücken zwischen Mathematik und Biologie: Mithilfe von Modellen berechnen sie die kleinsten Vorgänge in Zellen und Organismen
Bildquelle: istockphoto/NNehring
Ein wenig Licht genügt – und die Grünalge lässt sich als Produzent von Biokraftstoff nutzen. Damit dies in Zukunft effizienter gelingt, analysieren Mathematiker wie Alexander Bockmayr und Heike Siebert den Stoffwechsel des Mikroorganismus. Mit Formeln, Graphen und schnellen Computern bringen sie auch in weiteren Fällen die experimentelle Arbeit von Partnern aus Biologie und Medizin voran.
Das bestgehütete Geheimnis eines jeden Bäckers sind seine Zutaten: Nur wer die exakten Mengen zur rechten Zeit verrührt, erhält nach der Zeit im Ofen statt einem nassen Teig ein knuspriges Resultat. Reaktionen wie diese laufen in der Natur ganz ähnlich ab: Selbst kleinste Organismen verarbeiten bestimmte „Zutaten“ aus ihrer Umwelt zu neuen Stoffen – bei der Photosynthese etwa stellen Pflanzen aus Kohlendioxid und Wasser durch Lichtzufuhr Traubenzucker und Sauerstoff her. Für die Untersuchung derartiger biologischer Vorgänge interessieren sich Wissenschaftler in der mathematischen Systembiologie. Dabei sind die Ausgangsstoffe und Zielprodukte allerdings nicht so leicht zu überschauen wie beim Kuchenbacken.
Hinter dem Schreibtisch des Mathematik-Professors Alexander Bockmayr hängt darum ein großes Poster. Darauf sind unzählige Kreise und Quadrate durch Linien verbunden, die sich wiederum an bestimmten Punkten kreuzen. Es könnte der Schaltplan eines Atomkraftwerkes sein, tatsächlich handelt es sich aber um eine schematische Darstellung biochemischer Reaktionswege. Bereits in den 1970er Jahren haben Biologen die kleinsten Prozesse und Reaktionen in Lebewesen auf diese Weise veranschaulicht: „Wir haben es zu tun mit tausendenden von Molekülen und Reaktionen“, sagt Bockmayr, „mit einem komplexen metabolischen Netzwerk“. Der Wissenschaftler zieht gerne die Netzpläne des öffentlichen Nahverkehrs heran, wenn er Laien von seiner Arbeit berichtet. Vereinfacht betrachtet stehen Fahrgäste wie Mathematiker vor derselben Frage: Wie gelange ich mit bestimmten Linien über bestimmte Punkte an mein Ziel? Solche Bilder sind für Bockmayr ein wichtiges Instrument, um seine Disziplin zu erklären: Die mathematische Systembiologie ist eines der jüngsten Anwendungsgebiete der Mathematik, dessen Bedeutung in den vergangenen Jahren stark zugenommen hat.
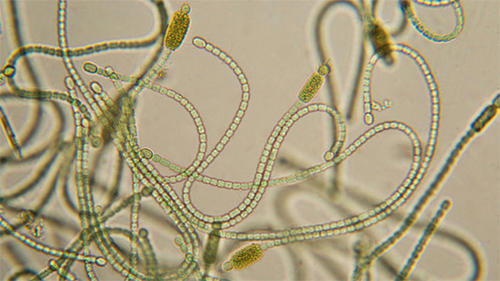
Der Stoffwechsel einzelliger Organismen steht derzeit bei zwei Forschungsprojekten von Bockmayr und seinem Team an der Freien Universität und dem DFG Forschungszentrum MATHEON im Mittelpunkt. In Zusammenarbeit mit Wissenschaftlern in Frankreich haben sie zum einen die Grünalge Chlamydomonas reinhardtii im Blick. In einem weiteren Projekt, das von der Einstein-Stiftung gefördert und gemeinsam mit Wissenschaftlern der Humboldt-Universität und der Hebräischen Universität Jerusalem durchgeführt wird, geht es um Cyanobakterien, auch Blaualgen genannt. So sehr sich Blau- und Grünalgen in ihren biologischen Eigenschaften unterscheiden, ihr Stoffwechsel lässt sich mit ähnlichen mathematischen Methoden untersuchen – und dies nicht nur um des Analysierens willen.
Beide Algenarten können nämlich zur Herstellung von Biokraftstoff benutzt werden. Aus Ausgangsstoffen wie Wasser oder Kohlendioxid lassen sich mithilfe der Algen etwa Ethanol oder Wasserstoff produzieren. Alles was die Algen für diese Reaktionen brauchen, ist ausreichend Licht. Bislang wird Ethanol oft aus Rohstoffen wie Mais gewonnen, die anderswo als Nahrungsmittel benötigt werden. Alternative Techniken sind also gefragt.
Wie an dieser Stelle die Mathematik ins Spiel kommt? Wer mit Algen möglichst viel Biokraftstoff produzieren will, muss die Stoffwechselwege kennen, auf denen er entsteht – am besten genau berechnet. Denn den Ausgangsstoffen steht im Netzwerk eine Vielzahl von möglichen Reaktionswegen offen. Von Interesse sind vor allem kurze Wege, Wege ohne viele Zwischenstationen. Von elementaren Stoffwechselpfaden spricht Bockmayr in diesem Zusammenhang. Obwohl es davon nur endlich viele gibt, ist die Anzahl noch immer sehr, sehr hoch: Bei großen Netzwerken liegen mehrere hundert Millionen Wege vor. „Selbst mit leistungsfähigsten Rechnern kann man das nicht einfach durchprobieren.“ Stattdessen entwickeln die Wissenschaftler mathematische Verfahren, die gezielt nur solche Pfade berechnen, die für die gegebene Fragestellung relevant sind.
Steht im Mittelpunkt der Forschung von Alexander Bockmayr: die Grünalge "Chlamydomonas reinhardtii".
Bildquelle: Alexander Bockmayr
Auch die richtigen Lichtbedingungen für ein möglichst großes Algenwachstum gehören zu den Variablen, die die Wissenschaftler untersuchen. Denn die Alge produziert nicht rund um die Uhr dieselben Stoffe. Wie der Mensch verhalten sich die Organismen bei Tag und Nacht völlig unterschiedlich. „Wenn die Sonne aufgeht, setzt das Licht eine Reihe von Prozessen in Gang“, sagt der Mathematiker, „das Bakterium beispielsweise will ja letztendlich wachsen und braucht dafür bestimmte Zellbausteine.“ Diese stellt es sich der Reihe nach bereit, während zeitgleich etliche weitere Vorgänge im Stoffwechsel ablaufen. „Man muss es sich so vorstellen, dass sich die Flüsse in einer bestimmten Reihenfolge auf das Netzwerk verteilen“, sagt Bockmayr. „Wie würden gerne verstehen, nach welchen Gesetzmäßigkeiten dies geschieht.“
Nicht nur der Algen-Stoffwechsel ist ein Fall für die Mathematiker der Freien Universität. Auch andere molekulare Netze werden in Dahlem erforscht: Signalwege etwa. Dabei werden zwischen Zellen bestimmte Moleküle übertragen, die die Zellentwicklung steuern. Sie lösen gewissermaßen die weitere Entwicklung der Zelle aus – dabei kann es sich schlicht um eine Teilung handeln, aber auch um den Beginn einer Erkrankung.
Der Ablauf im Körper ist dabei vergleichbar mit Dominosteinchen, die beim Umfallen eine Kettenreaktion auslösen: „Signalmoleküle kommen an einem Rezeptor an der Zellwand an und beeinflussen damit weitere Moleküle in der Zelle. Als Reaktion kann etwas Neues entstehen“, sagt die Mathematikerin Heike Siebert. Solche regulatorischen Netze und die Signalwege von Molekülen darin gehören zu den Forschungsschwerpunkten der Professorin. „Diese Prozesse sind unglaublich kompliziert, aber sie stecken überall.“
Von medizinischem Interesse ist dabei, wie man bestimmte Prozesse unterbinden und Reaktionen im Stoffwechsel ausschalten könnte. Siebert erforscht darum in den nächsten fünf Jahren mit ihrer Arbeitsgruppe und Kollegen der Charité – Universitätsmedizin Berlin beispielsweise die Vorgänge eines bestimmten Signalweges. Er ist bekannt dafür, dass er etwa den Zellzyklus und Zelltod steuert – und damit für die Krebsforschung relevant.
Medizinische Wirkstoffe könnten unerwünschte Reaktionen unterbinden helfen, indem sie den Signalweg beeinflussen oder ganz sperren. „Bislang zeigen erst Arzneimittelstudien, was bei der Blockade eines Signalweges passiert“, sagt Siebert. Mit mathematischen Modellen könne man jedoch bereits im Voraus herausfinden, welche Konsequenzen es für das Netzwerk hat, wenn bestimmte Verbindungen nicht mehr zustande kommen.
Bevor Bockmayr und Siebert solche Modelle am Computer entstehen lassen, ist oft monatelange Vorarbeit nötig. Unerlässlich dafür ist ein sehr enger Austausch mit den Kollegen aus der Biologie: Sie liefern die Anhaltspunkte dafür, wie die kleinsten Organismen „ticken“. Die Mathematiker suchen dann nach grundlegenden Zusammenhängen und Abhängigkeiten, um das biologische System auf ihre Disziplin zu übertragen – dieser Schritt gilt als schwierigster Teil der Arbeit. Mathematische Fragen wie diese bearbeitet Heike Siebert zunächst ganz klassisch mit Schmierpapier und Bleistift. Ihr früheres Biologie-Studium kommt ihr dabei zugute. Voraussetzung für die mathematische Systembiologie ist dieses Fachwissen nicht. Doch die Bereitschaft, sich in biologische Zusammenhänge einzuarbeiten, vereinfacht die Kommunikation mit den Partnern und damit die interdisziplinäre Zusammenarbeit. Daher legt Heike Siebert den Fokus ihrer Arbeit auf die Entwicklung der mathematischen Methoden. Der Gedanke an die spätere Anwendung sei zunächst einmal zweitrangig. „Die Mathematik ist universell: Ich kann verschiedenste Signalwege in verschiedenen Organismen mit denselben Methoden betrachten – unabhängig von der Interpretation im realen Kontext.“ Bei Alexander Bockmayr sind wiederum andere Vorerfahrungen nützlich. Vor seinem Wechsel an die Freie Universität im Jahr 2004 befasste er sich als Mathematiker mit der Produktionsplanung von Autos. „Ich stelle viele Analogien zur jetzigen Arbeit fest“, sagt der Professor, „denn auch die Zelle muss mit ihren Ressourcen effizient ein bestimmtes Produkt herstellen.“
Ist die mathematische Grundlage gelegt, folgt die eigentliche Entwicklung des Modells. Dabei bilden die Forscher die biologischen Prozesse digital nach: Sie zeichnen etwa die vorgegebenen Stoffwechselnetzwerke und weisen den einzelnen Komponenten bestimmte Eigenschaften oder Abhängigkeiten zu. Dabei beginnen sie auf recht einfacher Ebene und erweitern ihr Modell Schritt für Schritt um Details.
„Einerseits definieren wir dafür Graphen“, sagt Siebert. Diese geben Auskunft über die Struktur des Netzwerkes – dabei handelt es sich allerdings um ein statisches Bild. „Signale und ablaufende Reaktionen sind aber dynamische Prozesse“, erklärt Siebert. Solche Abläufe können etwa Differenzialgleichungen widerspiegeln. Diese Gleichungen können mehrere Variablen enthalten und beschreiben das Änderungsverhalten dieser Größen. „Man braucht aber sehr viele experimentelle Informationen, um die Gleichungen sinnvoll aufstellen zu können.“ In vielen Fällen reicht die in der Biologie vorliegende Datengrundlage jedoch nicht aus. Denn bestimmte Vorgänge auf der Mikroebene lassen sich in bislang möglichen Experimenten gar nicht messen.
Daher nutzen die Mathematiker abstraktere Modelle – die Rede ist dann von diskreter Modellierung. „Wir gehen davon aus, dass bestimmte Komponenten des Netzwerkes nur aktiv oder inaktiv sein können – auch wenn das in der Wirklichkeit natürlich nicht so ist“, sagt Siebert. Auch zeitliche Abläufe und die räumliche Verteilung lassen die Mathematiker außen vor, obwohl Informationen darüber vorliegen. „Es ist immer ein Kompromiss: Nutzt man zu viele Details, wird das Modell zu komplex und es ist nicht mehr zu handhaben“, sagt Siebert. Sie beschreibt die Modelle als Methode der Annäherung, mithilfe derer man die Dynamik verstehen könne.
Schließlich nutzen die Mathematiker die Modelle, um sich etwa Wege mit bestimmten biochemischen Eigenschaften ausgeben zu lassen – beispielsweise mit welchen Lichtbedingungen der Ethanol-Ertrag am größten ausfällt. Aber auch die Ergebnisse aus dem Modell sind meist so komplex, dass sie einer weiteren Analyse bedürfen – etwa im Vergleich mit Modellen anderer Gruppen für dasselbe Netzwerk. Bei der Modellbildung wie auch bei der Analyse sieht Siebert den Brückenschlag zur Biologie als notwendig an: „Wenn man bei einem Modell nicht genau versteht, was man weggelassen oder abstrahiert hat, ist es eigentlich wertlos.“
Mathematik und Biologie ergänzen sich, davon ist auch Bockmayr überzeugt. Das theoretische Arbeiten auf der einen und das experimentelle Forschen auf der anderen Seite ermögliche erst die Annäherung an bestimmte Probleme. Mithilfe der Mathematik können Biologen beispielsweise eine Vielzahl von Hypothesen auf einige wenige reduzieren. Nur mittels Experimenten wären manche Vorhaben ungleich aufwendiger oder gar nicht erst zu realisieren. Im Fall der Grünalgen analysierten die Mathematiker am Rechner die Flussverteilungen bei rund 100 verschiedenen Beleuchtungsbedingungen. 15 elementare Stoffwechselpfade waren für die ursprüngliche Fragestellung relevant: Mit dieser handlichen Auswahl können sich die Biologen im Labor der weiteren Untersuchung widmen.
Angesichts der vielen bereits entschlüsselten Prozesse auf molekularer Ebene glaubt Bockmayr an einen langsamen Wandel der Biologie: „Wir stellen uns vor, dass wir einen immer größeren Teil der Netzwerke mathematisch analysieren und beschreiben können.“ Ein Stück weit würden biologische Abläufe damit vorhersehbarer. Ähnlich wie es in der Physik ganz selbstverständlich mit mathematischen Methoden möglich ist, die Flugbahn einer Rakete bereits vor deren Start zu berechnen.